驼类动物体内含有3种不同亚型的抗体,分别是常规抗体IgG1、天然缺失轻链及CH1恒定域的重链抗体(heavy-chain antibody,HCAb)IgG2和IgG3。这种保留了全部抗原结合能力的天然无轻链及CH1区的单域抗体因其质量小(约15 kDa,为IgG的十分之一),也被称为纳米抗体(nanobodies,Nbs)或VHH抗体(variable domain of the heavy chain of heavy-chain antibody)。与传统抗体片段如抗原结合片段(fragment of antigen binding,Fab)和单链抗体(single chain antibody fragment,scFv)相比,纳米抗体(nanobodies,Nbs)具有多个明显的优势,比如免疫原性弱、生产成本低、水溶性好、组织渗透性好、稳定性与亲和力较高等。正是这些优良特性使得纳米抗体(nanobodies,Nbs)在生物技术方面得到了非常广泛的应用。
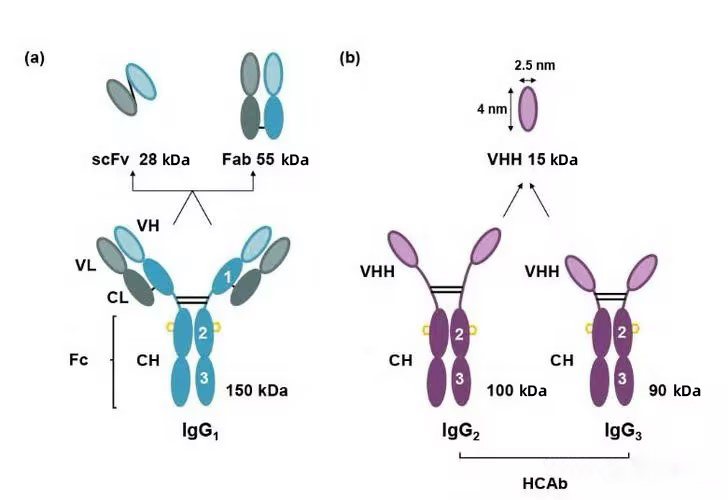
Structures and molecular mass of three different subtypes of antibodies and antibody fragments in camelid animals
对于大多数应用而言,稳定性是制约抗体应用的关键因素之一。一方面抗体在其生产、运输、储存及使用的过程中容易发生多种物理和化学降解,另一方面抗体的不稳定性聚集会潜在地影响产量、保质期和免疫原性等应用参数。因此抗体的稳定性不仅会影响其生物学和生化评估,还会影响纯化、储存以及配方设计和生产。在各种类型的抗体中,纳米抗体(nanobodies,Nbs)显示出极好的溶解性,并且对高温和化学变性具有显著的抗性,可以很大程度地克服传统抗体片段scFv的聚集和降解等稳定性问题,是许多应用的理想选择。
纳米抗体(nanobodies,Nbs)与常规抗体可变区(variable region of heavy chain,VH)的空间结构相似,其框架是由9个反向平行的β折叠片层(A-B-C-D-E-F-G-H-I)通过链间氢键和二硫键连接在一起组成。在此结构中,3个CDR分别连接BC、DE和HI链,并靠N端形成连续表面,与抗原表位的表面互补。连接CDR之间的氨基酸序列相对比较保守,称为骨架区(framework region,FR)。几乎所有纳米抗体(nanobodies,Nbs)结构都含有一个连接FR1(C23)和FR3(C104)的保守二硫键,该键跨越蛋白质的内部,将两个β链连接起来,增加了结构稳定性。部分纳米抗体(nanobodies,Nbs)还含有一个可限制CDR环柔韧性和构象自由度的额外二硫键。
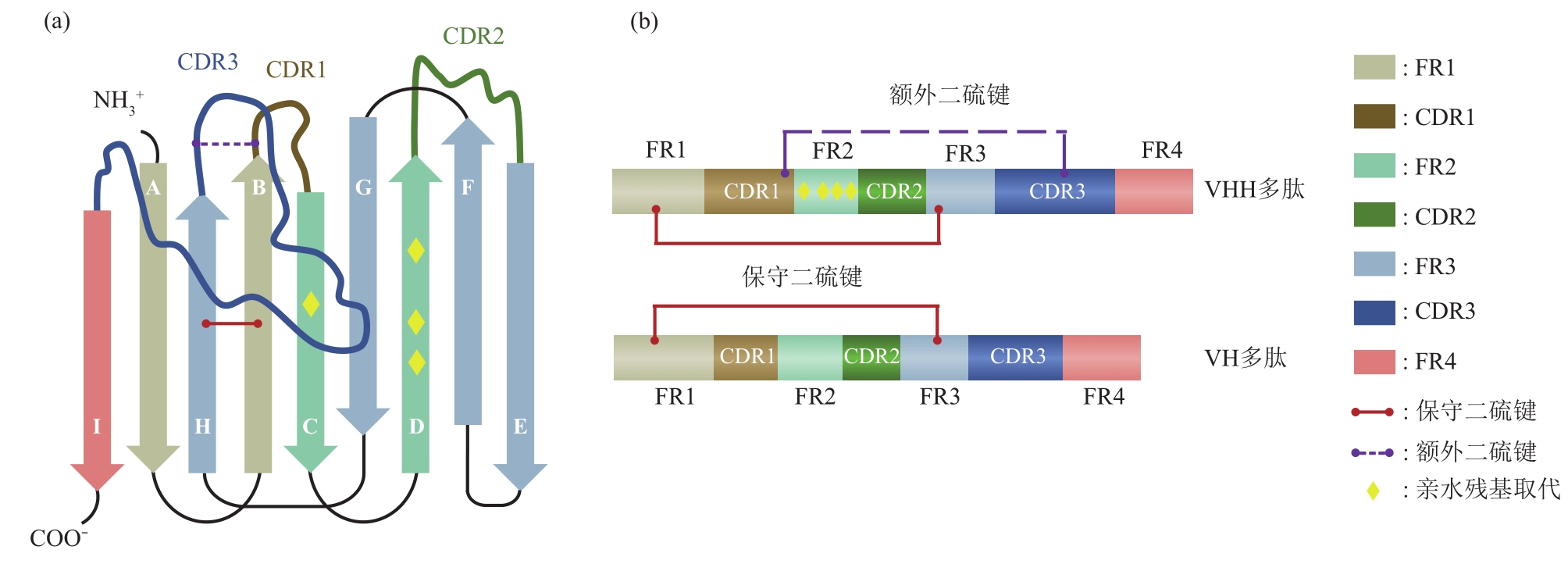 Structure of Nbs
Structure of Nbs
传统抗体VH和VL通过疏水作用力来稳定结构并共同构成抗原结合区。与之相比,纳米抗体(nanobodies,Nbs)仅含有3个可变区,尽管与抗原结合界面的表面积减少,但其仍具有较高的稳定性与亲和力。
它主要通过以下两种方式来适应轻链的缺失:
一、大量的亲水性氨基酸取代先前与CH1、VL结合界面的脂肪族残基(L12S、V42F/Y、G49E、L50R/C、W52G/L),且部分FR2被拉伸扭转的CDR3环覆盖,避免与外界水环境的接触,从而防止纳米抗体(nanobodies,Nbs)的二聚化,其CDR3环越长,纳米抗体(nanobodies,Nbs)越稳定。另外还可在CDR3末端形成疏水核心,有利于稳定Nbs的折叠结构域。
二、纳米抗体(nanobodies,Nbs)的CDR1和CDR3普遍比VH的长,潜在地增加了互补位构象的多样性,从而以高度的形状表面互补性与相应的抗原结合,一定程度上弥补了轻链缺失造成的抗原结合力下降以及因尺寸小而导致的潜在序列多样性降低。
纳米抗体(nanobodies,Nbs)的小分子和单域属性赋予了其比常规抗体更为稳定的特性,然而并非所有的纳米抗体(nanobodies,Nbs)都具有很好的热稳定性与化学稳定性。在以往研究的多个纳米抗体(nanobodies,Nbs)中,大约有2/3的纳米抗体(nanobodies,Nbs)在65℃处理后发生了不可逆的聚集,但是部分纳米抗体(nanobodies,Nbs)在90℃高温处理后仍具有90%的活性。这些稳定性差异的根本原因在于纳米抗体(nanobodies,Nbs)特殊结构的差异,包括氨基酸序列、二硫键的数量与位置、结构域的构象等。
那么,如何构建稳定性更高、亲和力更强的纳米抗体(nanobodies,Nbs)呢?普健生物深耕蛋白抗体领域15余年,在高质量纳米抗体(nanobodies,Nbs)制备技术上拥有自主核心技术,与知名科研单位、研究院所、医药研发企业成功合作相关项目20000+。在纳米抗体的制备方面,动物的选择是免疫成功的关键,普健生物自建养殖基地,每年近百头成年羊驼用于免疫,稳定提供大量的免疫抗原。通过设计目标抗原对健康强壮、精神良好、体型适中的羊驼进行四轮免疫后进行效价检测,当血清免疫效价达到预期后,再提取总RNA用于噬菌体文库的构建,利用噬菌体展示技术进行筛选得到高特异性和强亲和力的纳米抗体。
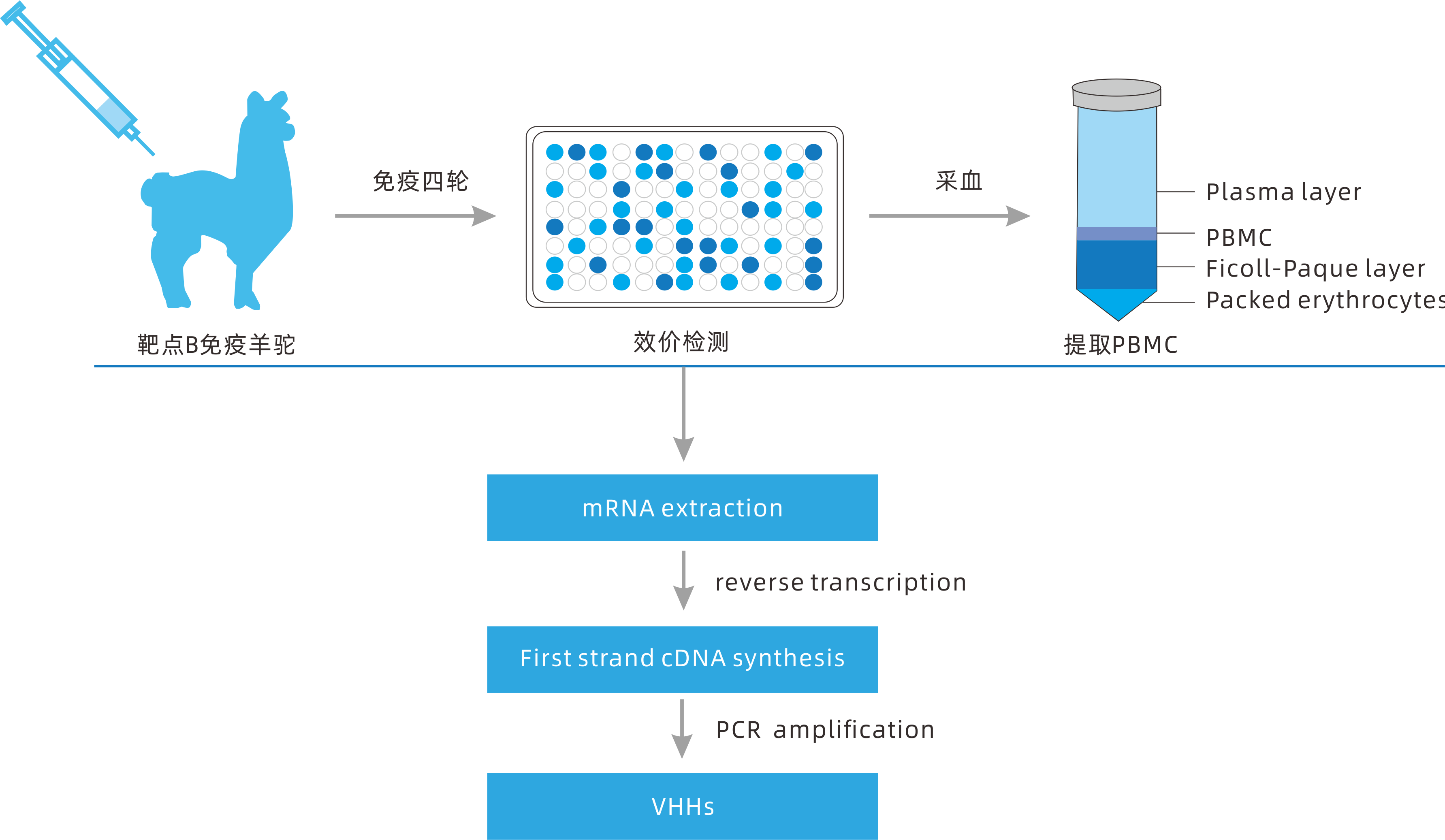
此外,普健生物还可根据科研需求,在分子设计时选择性加入功能区或基于纳米抗体(nanobodies,Nbs)结构特征采用合理的设计方法进行结构稳定性优化,例如共有序列驱动的序列修复、替换易于修饰的氨基酸、非天然二硫键的引入和CDR的移植。通过这些策略开发出在极端条件下仍能保持结合能力的稳定Nbs,从而为生物技术、检测、诊断和治疗应用提供高性能的试剂。
选择普健生物纳米抗体(nanobodies,Nbs)的优势
普健生物可以提供针对特定靶点的特异性纳米抗体定制服务,包括抗原检测,羊驼免疫,采血,效价检测,纳米抗体噬菌体库构建及淘选,特异性高亲和力纳米抗体筛选,表达,检测一条龙服务。亲和力可以达到108~1010M级别。
自建养殖基地:每年近百头成年羊驼用于免疫,稳定提供大量的免疫抗原
库容大:近千亿级别天然库
淘选周期短:最快在2周内可完成针对各类靶点的特异性纳米抗体快速筛选
亲和力高:抗体亲和力可达10-9M 级别
样本来源丰富:免疫库源自百余只不同种类的驼类样本-羊驼(Alpaca )、骆驼(Camel)、美洲驼(Llama),提供更高抗体多样性
超高品质:插入正确率100%,序列正确率97%,随机选取200个克隆测序,均无重复序列
可构建免疫库,获得更高亲和力VHH序列
参考文献
[1]Hamers-Casterman C, Atarhouch T, Muyldermans S, et al. Naturally occurring antibodies devoid of light chains. Nature, 1993, 363(6428): 446-448
[2]Li C, Tang Z, Hu Z, et al. Natural single-domain antibody-nanobody: a novel concept in the antibody field. J Biomed Nanotechnol, 2018, 14(1): 1-19
[3]Steeland S, Vandenbroucke R E, Libert C. Nanobodies as therapeutics: big opportunities for small antibodies. Drug Discov Today, 2016, 21(7): 1076-1113
[4]He T, Zhu J, Nie Y, et al. Nanobody technology for mycotoxin detection in the field of food safety: current status and prospects. Toxins, 2018, 10(5): 180
[5]Hoey R J, Eom H, Horn J R. Structure and development of single domain antibodies as modules for therapeutics and diagnostics. Exp Biol Med, 2019, 244(17): 1568-1576
[6]Liu M, Li L, Jin D, et al. Nanobody–a versatile tool for cancer diagnosis and therapeutics. Wiley Interdiscip Rev Nanomed Nanobiotechnol, 2021, 13(4): e1697
[7]Kunz P, Zinner K, Mücke N, et al. The structural basis of nanobody unfolding reversibility and thermoresistance. Sci Rep, 2018, 8(1): 7934
[8]Akazawa-Ogawa Y, Takashima M, Lee Y H, et al. Heat-induced irreversible denaturation of the camelid single domain VHH antibody is governed by chemical modifications. J Biol Chem, 2014, 289(22): 15666-15679
[9]Hagihara Y, Mine S, Uegaki K. Stabilization of an immunoglobulin fold domain by an engineered disulfide bond at the buried hydrophobic region. J Biol Chem, 2007, 282(50): 36489-36495
[10]Turner K B, Liu J L, Zabetakis D, et al. Improving the biophysical properties of anti-ricin single-domain antibodies. Biotechnol Rep, 2015, 6: 27-35
[11]Zabetakis D, Olson M A, Anderson G P, et al. Evaluation of disulfide bond position to enhance the thermal stability of a highly stable single domain antibody. PLoS One, 2014, 9(12): e115405
[12]Melarkode V A, Shinada N K, Narwani T J, et al. Discrete analysis of camelid variable domains: sequences, structures, and in-silico structure prediction. PeerJ, 2020, 8: e8408
[13]Lefranc M P, Lefranc G. IMGT® and 30 years of immunoinformatics insight in antibody V and C domain structure and function. Antibodies, 2019, 8(2): 29
[14]Govaert J, Pellis M, Deschacht N, et al. Dual beneficial effect of interloop disulfide bond for single domain antibody fragments. J Biol Chem, 2012, 287(3): 1970-1979
[15]Vincke C, Muyldermans S. Introduction to heavy chain antibodies and derived nanobodies. Methods Mol Biol, 2012, 911: 15-26
[16]Kunz P, Ortale A, Mücke N, et al. Nanobody stability engineering by employing the Delta ΔTm shift; a comparison with apparent rate constants of heat-induced aggregation. Protein Eng Des Sel, 2019, 32(5): 241-249
[17]Mendoza M N, Jian M, King M T, et al. Role of a noncanonical disulfide bond in the stability, affinity, and flexibility of a VHH specific for the Listeria virulence factor InlB. Protein Sci, 2020, 29(4): 1004-1017

 联系我们
联系我们





